来源:中国兽医协会
各分支机构、全体会员:
高效、精准、快速的疫病诊断与健康管理是疫病防控的关键。基于高通量靶向测序(Targeted Next-Generation Sequencing, tNGS)的检测技术能够快速获取样品中的病原体序列信息,实现动物病原微生物甚至包括一些人兽共患病病原的检测鉴定、溯源分型和综合表征的研判等,是对现有动物疫病核酸检测的有益补充。但目前该检测技术尚未在兽医检测行业达成一致性、规范性的认识。
为了填补这一空白,中国兽医协会兽医实验室检测分会组织了众多的行业资深专家,经过深入研究和讨论,形成了《动物病原微生物高通量靶向测序技术(扩增法)规范专家共识》。本共识涵盖了tNGS在动物病原微生物检测的应用场景、实验室基本要求、引物组设计指导原则、试验操作流程、数据分析、结果描述及判定、测序报告等多个方面,希望通过不断优化和完善检测流程和应用策略,进一步指导动物病原微生物检测工作,以更好地保障兽医公共卫生安全,促进畜牧养殖行业持续稳定发展。
附件:
动物病原微生物高通量靶向测序技术(扩增法)规范专家共识(2025版)
中国兽医协会
2025年6月11日
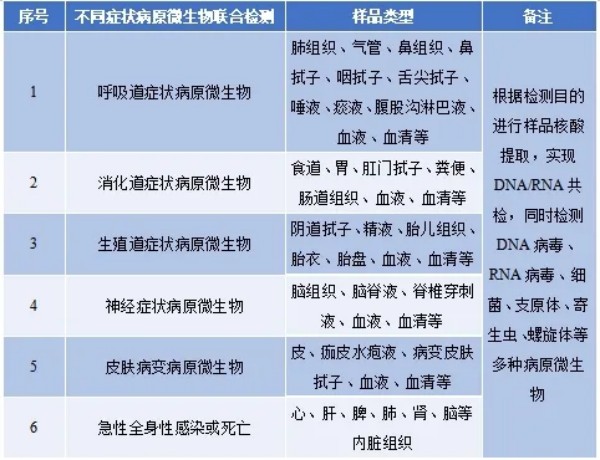
附件 动物病原微生物高通量靶向测序技术(扩增法)规范专家共识(2025版) 中国兽医协会兽医实验室检测分会 通讯作者: 田克恭 国家兽用药品工程技术研究中心,洛阳 471003; 王少林 中国农业大学,北京 100193 【摘要】基于高通量靶向测序(Targeted Next-Generation Sequencing, tNGS)的检测技术能够快速获取样品中的病原体序列信息,实现动物病原微生物甚至包括一些人兽共患病病原的检测鉴定、溯源分型和综合表征等,因此该项技术的发展既是对现有动物疫病核酸检测的有益补充,也是维护兽医公共卫生安全和畜牧养殖行业持续稳定发展的重要技术保障。由于目前尚未在兽医检测行业达成一致性、规范性的认识,本文将从tNGS在动物病原微生物检测的应用场景、实验室基本要求、引物组设计指导原则、试验操作流程、数据分析、结果描述及判定、测序报告等方面的专家共识进行阐述,并提出规范性要求和指导性建议。 【关键词】动物病原微生物;高通量靶向测序;扩增法;专家共识 养殖业是我国重要的经济组成部分,集约化、规模化、现代化养殖已然成为现在国内的主要养殖模式。然而,目前国内各大养殖场生产管理水平参差不齐,疫病流行情况复杂、病原种类繁多且多病原混合感染 频发、生物安全防控困难是目前制约畜禽健康养殖、降本增效的关键因素,而高效、精准、快速的疫病诊断与健康管理是疫病防控的关键。 此外,近年来公共卫生事件频繁发生,严重威胁人类健康和生态安全,“人病兽防、关口前移”是控制重大传染病传播的重要手段。动物病原微生物种类繁多,变异较快,畜禽、伴侣动物与野生动物可携带多种病原,且未知病原也不断出现。常规的PCR检测方法,单次检测病原数量有限,不能及时的发现病原变异的情况,无法识别未知病原、病原突变体以及混合感染等多种复杂情况。 tNGS通过靶向富集目标病原体或基因提高检测的敏感性与准确性,可分为多重PCR扩增法(简称扩增法)和探针捕获法[1]。其中多重PCR扩增法通过设计特异性引物进行超多重PCR富集目标病原体的靶核苷酸序列,而探针捕获法则通过探针杂交捕获的方式进行与探针法相比扩增法成本更低且速度更快,工作流程更简单,起始量要求更少,适于在兽医临床推广使用。基于扩增法的tNGS检测技术能够在短时间内快速获取样品病原微生物的详细序列信息,实现动物病原微生物甚至一些人兽共患病病原的检测鉴定、溯源分型、综合表征等,为疫病防控、诊断和治疗提供有力支持。 现在现阶段国内部分高校、兽医实验室建立了高通量测序平台,但是我国尚未出台动物病原微生物tNGS应用的规范性标准,并且在兽医行业还没有形成规范性、系统性、一致性的认知。为了满足兽医临床高通量靶向测序需要,本共识将对如何基于二代高通量测序平台开展动物病原微生物tNGS(扩增法)提出具体的指导性意见,并能将其应用于保障动物健康养殖和兽医公共卫生安全的解决方案,为提高动物疫病诊断、监测水平,保障养殖业持续稳定发展和公共卫生安全作出贡献。 1 tNGS在动物病原微生物检测中的应用场景 1.1 应用场景1 出现某种疾病的聚集性发病或暴发,怀疑是由微生物引起的感染性疾病,并且能根据临床症状推测疑似病原,且常规快速检测效率较低时,建议开展tNGS检测明确致病微生物。 1.2 应用场景2 病原微生物已明确或高度怀疑为病原菌,传统分菌及药敏试验操作无法最快速度或不能得到相关信息,可考虑使用测序方法进行病原菌的鉴定及耐药基因检测。由于宏基因组测序mNGS常规数据量对耐药基因检测存在局限性,随机测序的方案仅在高浓度时可稳定检出耐药基因,此时可考虑对样品进行耐药基因tNGS检测。需明确,耐药基因的检出情况与样品的细菌载量相关。 1.3 应用场景3 病原微生物已明确或高度怀疑为某病原微生物,且临床表现提示该病原微生物可能具有特殊的毒力表型,需要进一步确认是否携带重要毒力因子时,可以考虑对感染样品进行tNGS物种鉴定,同时检测毒力基因。 1.4 应用场景4 传统病原学检测的结果不能解释临床表现的全貌或抗感染治疗的反应,怀疑同时存在其他病原感染,或想进一步明确感染病原的亚型,建议在完善其他更多检测技术的同时,开展tNGS检测。 1.5 应用场景5 采集动物源细胞含量较高的样品,如肺泡灌洗液等;或采集污染较严重样品,如粪便等,如果需进行测序,tNGS敏感性会优于mNGS。 1.6 对于通过常规微生物学检查容易明确的病原微生物,且经验治疗通常有效的感染,不建议常规进行tNGS检测,也不建议应用tNGS来评估抗感染治疗效果[2]。 2 实验室基本要求 2.1 生物安全要求 高通量测序的动物种类和病原微生物种类范围广泛,可能存在高致病性病原微生物。因此,开展动物病原微生物高通量靶向测序的实验室应具备生物安全二级(Biosafety Level 2,BSL- 2)及以上的条件,并严格按照农业农村部要求及相关法律法规进行实验操作[3]。在样品检测完成后,确认无需对样品进行保留即按照实验室操作手册做无害化处理。 2.2 试验操作区布局要求 tNGS技术平台的试验操作主要包括试剂配制、样品接收与前处理、核酸提取、靶标区域扩增、测序文库构建、上机测序和数据分析等步骤。因此,原则上实验室应设置以下区域:样品接收区、试剂准备区、样品制备区、扩增区、文库构建区、质检区和测序区、数据分析及报告区[2][4]。 2.3 实验室自建方法基本要求 2.3.1 样品前处理方法 实验室应具备针对不同类型样品的前处理方法,无论DNA还是RNA核酸提取质量都应满足测序要求,并且在充分保障生物安全性的前提下,建立完整的核酸提取流程[5][6]。 2.3.2 实验室防污染策略 靶向测序可能的污染来源主要由是核酸提取、扩增及建库过程中引入的污染,建议实验室自建有效的防污染策略,包括使用阴性对照文库的方法、生物信息降噪措施处理[1]等。 2.3.3 自建数据库基本要求 建议实验室验证自建的数据库中病原基因组序列的准确性,过滤数据库错误和不完整的序列,同时定期更新病原基因组的数据库,以提高检测的敏感性和准确性。 2.3.4 方法验证基本要求 实验室建立检测方法后,要充分验证方法的准确性、敏感性、可重复性,并且建议对某些常见感染物种进行检出限、携带和交叉污染的验证。此外,对分析过程需完善分析的各项指标。包括分析敏感度(指能够通过测序获得准确目标微生物的最低浓度)、分析特异性(主要评价样品中潜在干扰物质对检测结果的影响)、不合格统计率等。 3 引物组设计的指导原则 3.1 靶标基因选择原则 病原微生物tNGS主要通过对目标病原微生物靶标基因的测序实现病原微生物的鉴定与其他特征分析。这些靶标基因包括根据病原微生物基因组特征选择“特异性鉴定基因”、“特异性毒力基因”“耐药性基因”以及“高突变基因”等其他功能基因,所有以上不同特征的基因都可称为病原微生物的靶标基因。 在利用靶标基因鉴定病原微生物时,需明确病原微生物与靶标基因之间的联系,原则上满足靶标基因仅对应单个病原微生物;对于具有功能表征的靶标基因,不仅要有助于鉴定,还应具有实际检测意义,如高毒力基因、高流行率耐药基因;对于具有突变监测作用的靶标基因也应在鉴定的同时具有监测意义,如疫苗蛋白编码基因。 病原微生物靶标基因的组合应灵活,但并非覆盖的种类并非越多越好,需充分考虑病原微生物本身特点来选择合适的靶标基因组合,避免测序数据分配不均,出现假阴性结果。 3.2 靶标及引物之间最低干扰原则 第一,需保证病原靶标基因之间同源性较低或无同源性;第二,需保证引物之间同源性较低或无同源性。若第一点无法满足,则在设计引物时必须满足第二点,确保引物之间不存在交叉。 3.3 引物长度控制原则 每对引物的扩增长度需控制在200 bp以内,以100-200 bp为宜,以达到能够高效、特异地对靶标基因进行靶向扩增为最终目的。 3.4 靶点区域选择原则 对于保守鉴定基因,需选择高特异性区域为引物扩增区;对于突变基因,则需选择高突变区域为引物扩增区。各个靶标基因设计的引物对数应根据需要覆盖区域进行调整。 4 试验操作流程 按照各测序仪器厂商提供的测序样品制备流程和测序操作进行测序样品的处理、文库构建和测序。基本流程见图1。 图1 实验基本流程 4.1 样品采集与运输 动物病原微生物tNGS可根据动物的临床症状和检测目的选择合适的测序样品,原则上,应优先采集与检测目的直接相关的病料,临床取材部位应选择病变组织与健康组织交界部位的样品,样品选择示例如表1所示。 表1 样品选择示例 tNGS检测的样品采集应严格按照无菌要求执行,使用一次性无菌采样袋或者采样管等收集样品,以防止交叉污染。每个动物的样品都应单独保存和编号,并注明样品的来源、采样日期、患病动物的临床症状、目标病原以及采集人员的基本信息等。采集后的样品若不能及时送检,请于低温条件(4℃或-20℃)保存,并在送检时采用冷链运输,以防止样品在运输过程中降解。样品的采集与送测过程都应遵循国家生物安全样本的采集与转运要求[6]。送检人员应指明需要特别关注的病原微生物。 4.2 样品前处理 样品前处理的主要目的是从各种类型的送检样品中提取微生物核酸以确保后续测序步骤的有效进行,应根据不同测序平台说明书要求进行样品微生物核酸的质量检测与定量分析。 在进行样品前处理时,应遵守从洁净到污染、从简单到复杂的规则,优先处理全血、组织等相对干净的样品,再处理拭子类、粪便等污染性样品。保证同一类样品同一处理,不同类样品不交叉处理,避免样品之间交叉污染。 4.2.1 核酸提取 核酸提取效率在不同微生物间存在差异,通常提取难度表现为真菌>革兰阳性菌>革兰阴性菌>病毒,提取方法主要分为溶液型抽提、柱式抽提和磁珠纯化法三种,每种方法对提取微生物的DNA/RNA各有侧重。若怀疑细菌、真菌、DNA病毒、寄生虫等病原微生物感染且需进行高通量靶向测序检测时,建议采用DNA策略;若怀疑RNA病毒感染时,建议采用RNA策略;若怀疑RNA病毒与其他类型病原微生物混合感染时,则建议采用DNA+RNA策略[7][8]。 ① DNA策略:提取真菌、细菌、寄生虫以及DNA病毒等病原的DNA。 ② RNA策略:提取真菌、细菌、寄生虫以及RNA病毒等病原的RNA,并反转录为cDNA。 ③ DNA+RNA策略:提取真菌、细菌、寄生虫以及DNA病毒等病原的DNA以及真菌、细菌、寄生虫以及RNA病毒等病原的RNA,在不去除gDNA的同时,反转录为cDNA。 实验室应充分评估各提取方法或试剂性能,并根据不同样品类型和测序目的选择最合适的提取方法或试剂。 4.2.2 核酸质量评估 提取核酸后,应对其质量进行验证,验证指标主要包括核酸的浓度。通常,靶向测序对于核酸质量的要求相对较低。在不要求核酸完整性的前提下,建议充分裂解细胞,确保所有微生物核酸能最大程度裂解释放。 应使用合适的DNA基因组和文库构建试剂进行文库构建,DNA样品的类别选择标准应参照表2,RNA样品的类别选择应参照表3。 表2 进行文库构建的DNA样品类别选择标准 表3 进行文库构建的RNA样品类别选择标准 4.3 测序文库构建 tNGS文库构建是使用特异性引物池富集目标区域的DNA分子,根据不同检测目的,将不同动物病原微生物进行适当组合,可以在明确病原谱的前提下富集目标区域的待测DNA分子,并将其连上特定的检测接头序列,包括分子标签等。选择适合不同测序平台的文库构建方法,可以有效避免上机样品出现较大偏倚,从而尽可能地减少测序误差。 在构建测序文库的过程中,需同时建立阴性对照与阳性对照文库,以控制建库过程中可能引入的污染。 4.3.1. 阴性对照文库 (1)实验室背景 在建库过程中,应使用无核酸水、buffer液、细胞液等病原空白物质来构建阴性对照文库,以排除实验室背景污染。 (2)外参核酸spike-in 若靶向扩增panel已设计内参,为最大限度保证样品病原数据量不受其他因素影响,无需添加外参spike-in;若靶向扩增panel未设计内参,为保证低质量核酸,如C类核酸,能成功上机测序,可考虑添加外参spike-in。 4.3.2 阳性对照文库 选择已知病原类型物质,包括病原核酸标准物质、病原靶标基因质粒、病毒株或菌株等,构建阳性对照文库。 4.4 待测文库质量控制 4.4.1 扩增子文库质量评估 为保证文库能正常上机,且数据量满足分析要求,一般建议单个扩增子文库的浓度≧ 5 ng/µL,而且扩增子条带的电泳图为单一条带,条带大小(bp)=扩增子目标片段大小(bp)+标签片段大小(bp)[+外参片段(bp)]。 4.4.2 上机文库质量评估 为保证文库能正常上机测序,且下机数据量达到分析要求,应根据各自测序平台的要求对上机文库的质量进行评估,如果上机文库的质量达到质检标准,则可以进行测序分析,否则需重新制备文库。 4.5 测序 应使用适合的高通量测序试剂盒,按照测序仪厂商提供的标准测序流程进行高通量测序操作。 5 数据分析 生物信息分析是对测序后的原始序列数据进行分析和处理,对于靶向测序的检测结果具有重要的决定性意义。高通量靶向测序的基本生物信息分析流程包括数据的质量控制与过滤、比对与分析等[9]。 5.1 质控步骤 tNGS的样品原始数据,应首先进行质量控制,包括去除含接头和低质量的序列,然后才能进行后续的生物信息学分析。 在解读报告时,应考虑以下几个参数,包括测序质量(包括是否去除低复杂度和低质量序列,以及Q20、Q30等指标)、覆盖率、测序深度、以及微生物序列的数据量等。 测序完成后,需要对Q20和Q30的指标进行统计和评估。对于每个检测样品,可用数据对应的碱基识别质量值进行统计判定,应符合如下要求: ——大于Q20的碱基比例≥90%; ——大于Q30的碱基比例≥80%。 5.2 可用数据量要求 为达到数据分析要求,每批样品上机时分配到单个样品的原始数据量不低于3 M,且可用的测序数据量应足以证明从样品中检测到所声明的靶标病原物种。此外,每个样品测序生成的可用高质量序列数目(包括单端测序的序列条数,或双端测序的序列对数)应大于1×104条。 5.3 靶标基因序列比对分析 目前国内尚无专门化动物病原微生物基因序列数据库,搭建的病原微生物靶标基因数据库的基因数据一般来自公开数据库或文献中的数据库,比如NCBI GeneBank、NCBI RefSeq、国家微生物科学数据中心发布的gcPathogen等。 使用fastq进行数据质控后,参考病原靶标基因数据库,用pblat软件和定制化生信脚本,对样品数据进行序列对比分析,内参靶标基因检出率不低于10%,则测序试验数据可靠。样品病原靶标基因覆盖序列的读长不小于测序长度的80%(如测序读长150 bp,则获得的靶标基因序列不少于120 bp),序列的同源性不低于95%,E值小于1e-20。 6 结果描述及判定 建立tNGS病原微生物检测的阈值影响因素较多,包括测序平台、测序流程、样品类型、病原种类、患病动物状况等等,为防止不同实验室背景干扰造成的假阳性结果,建议各实验室在开展检测时建立基线数据库,包括阴性对照基线数据库和历史检测基线数据库来提高检测结果的准确性[2]。综合样品类型、建库方法与基线数据库情况来看,各类病原检出的敏感性高低顺序依次为细菌/支原体、DNA病毒、RNA病毒。因此,各实验室应在本实验室的基线数据库的标准下充分考虑不同类型病原检出的特点,再设定相应判定的阈值。但需注意,不同样品tNGS检测的reads数不具有可比性,不能简单依靠reads数判断动物感染状态。 基于批量检测样品提出以下经验性判定标准,可供参考,各实验室应结合建库方法、仪器平台、设备基线数据库等参数对不同类型病原判定阈值进行调整。 6.1 阳性 比对分析后,若所有病原靶标基因序列数目不少于1000条,且特定病原靶标基因序列数目不少于100条,同时在阴性对照文库中无检出,可以判定该病原靶标基因有检出,该病原检测结果为阳性。 6.2 阴性 比对分析后,若所有病原靶标基因序列数目不少于1000条,但特定病原靶标基因序列未检出或数目少于20条(参考实验室的基线数据库),同时在阴性对照文库中无检出,可以判定该病原靶标基因无检出,该病原检测结果为阴性。但阴性结果不建议单独作为排除依据,应结合tNGS的靶标覆盖范围、样品类型和其他临床信息综合判断。 6.3 可疑 比对分析后,若所有病原靶标基因序列数目不少于1000条,且特定病原靶标基因序列数目在20-100条之间,同时在阴性对照文库中无检出,则判定该病原靶标基因检出结果为可疑,建议对该样品进行重复试验。 6.4 重要病原 当tNGS检出高传染性或高致病性病原微生物,以及重要人兽共患病原微生物时,无论特异性序列数量为多少,都应给予充分重视,需对检测结果进行复核,并使用其他检测技术进行验证[10]。 7 测序报告 7.1 报告内容 报告单应详细列出以下关键信息:动物类型、采样与收样日期、送样单位名称、联系人及联系方式、检测人员姓名及联系方式、送检样本类型与数量、患病动物临床表现、检测包含的病原微生物中文名称及拉丁文名称、检出的病原微生物的中文名称及拉丁文名称、检出病原微生物的丰度以及病原感染情况等。同时,报告中应提供对专业术语和技术参数的注释。 7.2 报告时间 报告时间应在48~72小时内完成并发放。 7.3 报告解读 tNGS病原微生物检测结果,建议由具有一定生物信息学知识和兽医专业背景的人员进行解读与审核。在解读过程中,需要结合患病动物的临床表现以及同一养殖区域内其他动物表现等因素进行综合判断。若不进行正确解读与甄别,盲目依据tNGS报告开展治疗,可能会导致抗微生物药物的滥用[6]。 7.4 阴性结果报告 tNGS结果为阴性,对于排除感染常具有较好的阴性预测值。但应充分考虑样品与引物间的数据量分配、病原微生物载量及核酸提取程度、不同实验室基线数据库标准等客观因素,必要时建议进行二次测序检测或对疑似病原微生物进行其他检测方法检测。 8 小结 tNGS技术是从核酸中靶向富集目标病原体序列,可实现病原与重要耐药基因、毒力基因的快速精准检测。该技术在人医临床诊断如混合感染、未知病原感染以及综合症检测等领域具有显著的优势,能够快速鉴别感染病原微生物的种类,对病原感染载量进行定量,并对特定物种的耐药基因进行分析,从而精准实现重要病原的全面覆盖,并提升整体的诊断速度。该技术可适用于动物疫病检测-监测-预警的整体链条环节。本共识首次提出tNGS在动物病原微生物检测中的应用规范,希望通过不断优化和完善检测流程和应用策略,进一步指导动物病原微生物检测行业,以更好地保障兽医公共卫生安全,促进畜牧养殖行业持续稳定发展。 参与编写本共识的人员及单位 王少林 中国农业大学 原 霖 北京中科基因技术股份有限公司 张桂红 华南农业大学 顾小雪 中国动物疫病预防控制中心 赵灵燕 浙江省动物疫病预防控制中心 赵东明 中国农业科学院哈尔滨兽医研究所 卢受昇 广东省动物疫病预防控制中心 孙彦阔 华南农业大学 李瑞超 扬州大学 冯小宇 北京市动物疫病预防控制中心 卫秀余 上海市奉贤兽医研究所 唐 岳 深圳华大智造科技股份有限公司 赵永旭 法国诗华动物保健公司 田克恭 国家兽用药品工程技术研究中心 左 扬 中国农业大学 参考文献 [1] 中国医疗保健国际交流促进会临床微生物学分会. 靶向高通量测序在感染性疾病中应用与实践专家共识[J]. 中华医学杂志, 2024, 104(48): 4375-4384. [2] 中华医学会儿科学分会新生儿学组,中华儿科杂志编辑委员会. 宏基因组二代测序技术在新生儿感染性疾病中的临床应用专家共识[J]. 中华儿科杂志, 2022, 60(6): 516-521. [3] 中国兽医协会兽医实验室检测分会. 动物病原微生物宏基因组高通量测序技术规范专家共识(2024版). 中国兽医协会, 2024. [4] 中华医学会检验医学分会. 宏基因组测序病原微生物检测生物信息学分析规范化管理专家共识[J]. 中华检验医学杂志, 2021, 44(9): 799-807. [5] 中华医学会检验医学分会临床微生物学组,中华医学会微生物学与免疫学分会临床微生物学组,中国医疗保健国际交流促进会临床微生物与感染分会. 宏基因组高通量测序技术应用于感染性疾病病原检测中国专家共识[J]. 中华检验医学杂志, 2021, 44(2): 107-120. [6] 中华医学会检验医学分会. 高通量宏基因组测序技术检测病原微生物的临床应用规范化专家共识[J]. 中华检验医学杂志, 2020, 43(12): 1181-1195. [7] 中华医学会神经病学分会感染性疾病与脑脊液细胞学学组. 中枢神经系统感染性疾病的脑脊液宏基因组学第二代测序应用专家共识[J]. 中华神经科杂志, 2021, 54(12): 1234-1240. DNA/RNA. [8] 《中华传染病杂志》编辑委员会. 中国宏基因组学第二代测序技术检测感染病原体的临床应用专家共识[J]. 中华传染病杂志, 2020, 38(11): 681-689. [9] 宏基因组分析和诊断技术在急危重症感染应用专家共识组. 宏基因组分析和诊断技术在急危重症感染应用的专家共识[J]. 中华急诊医学杂志, 2019, 28(2): 151-155. [10] 中华医学会细菌感染与耐药防治分会. 呼吸系统感染中宏基因组测序技术临床应用与结果解读专家共识[J]. 中华临床感染病杂志, 2022, 15(2): 90-102.